Help
TogoVar全般
利用条件に関して
TogoVar収録データに関して
TogoVar検索システムに関して
TogoVar全般
TogoVarの目的や有用性は何でしょうか。
ゲノムの解読はNGSにより低頻度の変異を取得することが可能となりました。しかし、欧米の既存データベースには、十分なサンプル数の日本人集団における頻度データはありません。この問題を解決するため、公開されている日本人のゲノム変異情報やNBDCに寄託されたヒトデータベースからの情報を集積し、より信頼性の高い頻度情報を提供することを目指します。
また、ヒトゲノムの参照配列は常に一定ではなく更新される情報であるため、同じバリアントであっても参照配列が異なるとゲノム上の位置が異なります。そこで、当データベースでは同じバリアントにはユニークなIDを付与することで、研究者が行っているバリアントの位置情報を参照配列間で変換する作業を省力化します。
利用条件に関して
TogoVarデータベースの利用は無償でしょうか。
すべて無償での利用が可能です。データベースの利用については、Termsのページを参照してください。
TogoVarデータベースを論文で引用するにはどうすれば良いでしょうか。
Termsのクレジットの表示例を参考にしてください。
商用利用は可能でしょうか。
TogoVarのwebサイトの閲覧や公開予定のdockerイメージは商用利用可能です。TogoVarが取り込んでいる第三者作成データに関してはそれそれの利用条件に従ってください。詳しくはTermsをご覧ください。
TogoVar収録データに関して
個人レベルの表現型情報の取得は可能でしょうか。
当データベースは個人情報を保持しておらず、また、個人ごとの表現型情報は現在のところ提供はしておりません。表現型情報の詳細については、JGA-WESデータセットおよびJGA-SNPデータセットに記載されている研究グループに直接お問い合わせ下さい。
遺伝子発現やタンパク質の情報とは結び付かないのでしょうか。
当データベースはヒトゲノムの遺伝子変異について、日本人の公開情報を中心として統合したものです。当データベースはRDFの技術をベースとして利用している為、遺伝子発現やタンパク質の情報がRDFを利用しているデータベースである場合は、容易に拡張が可能と考えております。遺伝子発現やタンパク質の情報への拡張は順次実施する予定です。
バリアントの検出の際に使用したソフトウェアおよび品質管理の方法を教えてください。
TogoVarデータセット作成方法を参照ください。
TogoVarで対象としているバリアントの種類は何ですか。
SNV、Insertion、Deletion、Indel、Substitutionの5種類です。HGVSがType of variantsに列挙した5種類のうち、DuplicationはInsertionに、DeletionとInsertionの長さが同じIndelはSubstitionに分類されています。なお、バリアントの種類の特定には、Variant Effect Predictor (VEP)を利用しています。
Consequenceの値はどのように計算していますか。
Variant Effect Predictor (VEP)のvariant consequenceの値を表示しています。1バリアントに対応するトランスクリプトが複数ある場合は、一番重大なconsequenceのみを表示している場合があります。
Clinical Significanceの値はどのように計算していますか。
AlphaMissense、SIFT、PolyPhenとは何ですか。
AlphaMissense、SIFTおよびPolyPhenはアミノ酸配列が変更された際のタンパク質の機能への影響を予測します。これらのプログラムはVariant Effect Predictor (VEP)を使用して計算しました。以下のアイコンは定性的予測に使用される閾値スコアを表しています。
- AlphaMissense> 0.564Likely pathogenic≥ 0.340Ambiguous「Likely pathogenic(おそらく病原性あり)」と 「Likely benign"(おそらく良性)」のスコアの閾値は、それぞれ病原性および良性のClinVarバリアントを90%の精度で分類できるように選ばれました。詳細はAlphaMissense in VEP Pluginsを参照ください。< 0.340Likely benign
- SIFT< 0.05Deleteriousこのスコアはアミノ酸の置換が許容される確率を正規化して示したものです。スコアがゼロに近い場合、その置換が有害である可能性が高いと考えられます。このスコアに基づいて定性的な予測が行われます。0.05未満のスコアを持つ置換は「有害 (Deleterious)」と分類され、それ以外のスコアは「容認 (Tolerated)」とされます。詳細はSIFT in Pathogenicity prediction of Ensembl Variationを参照ください。≥ 0.05Tolerated
- PolyPhen> 0.908Probably Damaging> 0.446Possibly Damaging≤ 0.446Benign推定偽陽性率(FPR)が5%以下の事後確率スコアを持つバリアントは、「Probably Damaging (おそらく有害)」と予測されます。FPRが10%以下の場合は「Possible Damaging (有害の可能性あり)」とされ、FPRが10%を超えるバリアントは「良性 (Benign)」と分類されます。詳細はPolyphen in Pathogenicity prediction of Ensembl Variationを参照ください。UnknownUnknown
バリアントデータ作成時にリフトオーバツールを利用しましたか。
はい。一部のGRCh38データセットは、そのGRCh37データセットをリフトオーバして作成しました。対象データセットはTogoVar収録データセット一覧 (GRCh38)のLiftover GRCh37を参照ください。リフトオーバツールはtransannoを利用しました。2023/7/14リリースまではCrossMapを利用しました。
TogoVarの検索システムに関して
検索結果のFrequency列に表示される頻度メーターは何を表現していますか。
各データセットにおけるAlternative allele頻度を表現しています。頻度メータの色はデータセットに割り当てられた色に対応しています。データの有無や頻度の値を9段階で表現しています。
バリアントタイプごとにPosition、Ref、Altはそれぞれどのように記述されますか。
以下に、各バリアントタイプ毎の例を示します。染色体の最初の塩基の位置は1です。
Variant Type | ID | Position | Ref / Alt | 説明 |
|---|---|---|---|---|
SNV | tgv167913215 | 12 111767141 | CA | 12番染色体111767141番目の塩基CがAに置換された |
Ins | tgv134135934 | 12 111786238 | G | 12番染色体111786238番目と111786239番目の塩基の間に塩基Gが挿入された |
Del | tgv110307325 | 12 111778836 | A | 12番染色体111778836番目の塩基Aが欠失した |
TogoVarでは、どのようにバリアントを表記していますか。
TogoVarでは、ゲノム上の位置及びreference allele、alternative allele、ヒトゲノム標準配列のビルド番号の組み合わせでバリアントを一意に識別します。TogoVar及びdbSNP、VCF、HGVSでの表記例を下の表に示しています。
TogoVar | dbSNP | VCF | HGVS | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
ID | Position (GRCh38) | Ref / Alt | ID | Position (GRCh38) | Alleles | Position | Ref | Alt | ||||||
SNV | tgv167913215 | 12 111767141 | CA | rs567143183 | chr12:111767141 | C>A | 12:111767141 | C | A | 12:g.111767141:C>A | ||||
| tgv47263142 | CG | C>G | C | G | 12:g.111767141:C>G | |||||||||
Ins | tgv134135934 | 12 111786238 | G | rs534925026 | chr12:111786238 | insG | 12:111786238 | T | TG | 12:g.111786238_111786239insG | ||||
Del | tgv110307325 | 12 111778836 | A | rs766411401 | chr12:111778836-111778847 | delA | 12:111778835 | GA | G | 12:g.111778847del | ||||
Human Genome Variation Society (HGVS) 表記(例:ALDH2:p.Glu504Lys) で検索できますか。
はい。HGVSc (例:ALDH2:c.1510G>A)とHGVSp (例:ALDH2:p.Glu504Lys)で検索可能です。Variant RecoderでHGVS表記をGRCh38またはGRCh37座標に変換します。
Advanced searchでできることは何でしょうか。
代替アレル頻度やClinical significanceなどの検索条件をANDやORで結合したクエリーを作成できます。使い方は、Q:日本人集団とその他の集団で代替アレル頻度が異なるバリアントを検索できますかをご覧ください。
日本人集団とその他の集団で代替アレル頻度が異なるバリアントを検索できますか。
はい。Advanced searchによる検索が可能です。
代替アレル頻度の比較対象のバリアントは以下の通りです。- 日本人集団はGEM-J WGAまたは8.3KJPNのいずれかに含まれるバリアント
- gnomADはgnomaAD genomesまたはexomesのいずれかに含まれるバリアント
- 画面上部のAdvanced searchタブを選択します。
- Add conditionメニューからAlternative allele frequency/countを選択します。
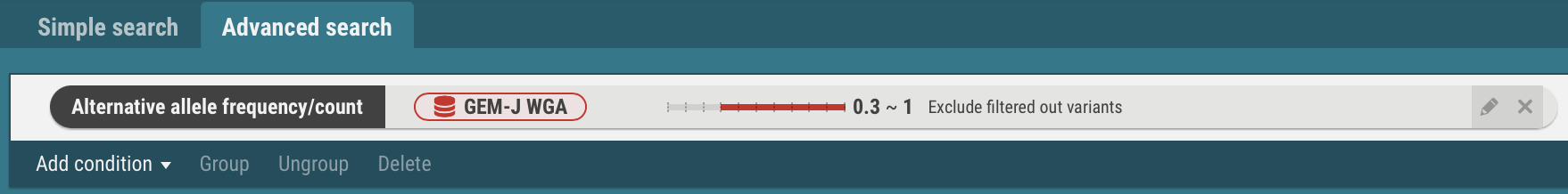
- 以下の画面でデータセット(GEM-J WGA)と代替アレル頻度(>=0.3)を指定します。代替アレル数でも指定できます。
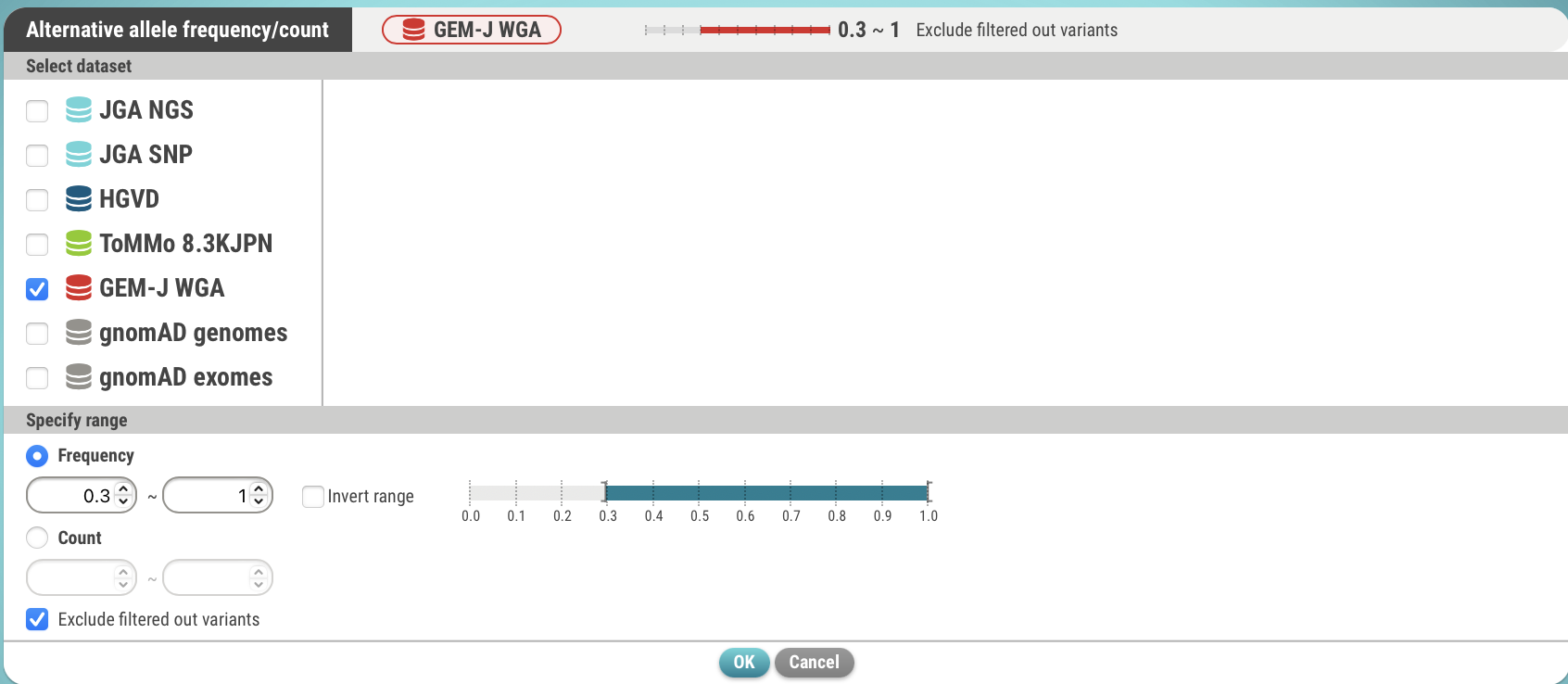
- OKボタンを押下します。検索条件が設定されました。
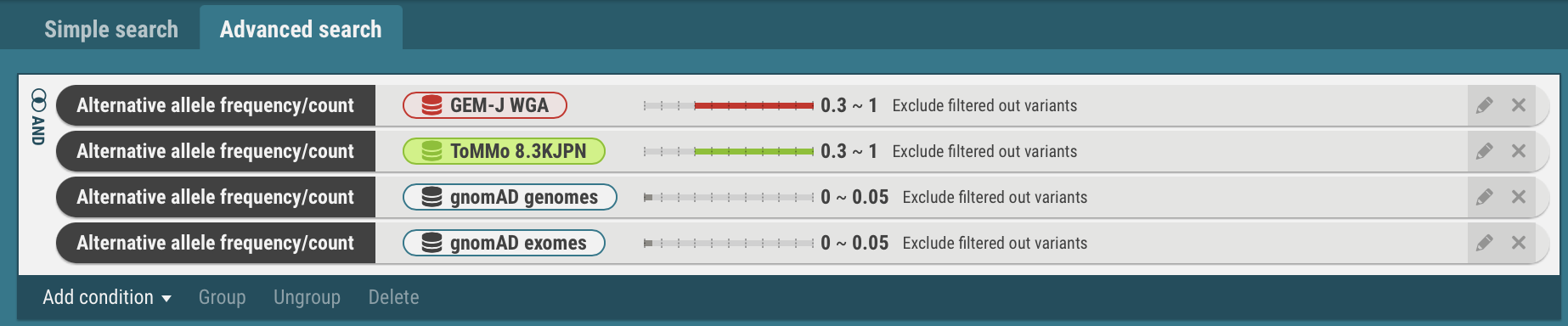
- 同様の手順でデータセットToMMo 8.3KJPN、gnomAD genomes、gnomAD exomesについても検索条件を設定します。
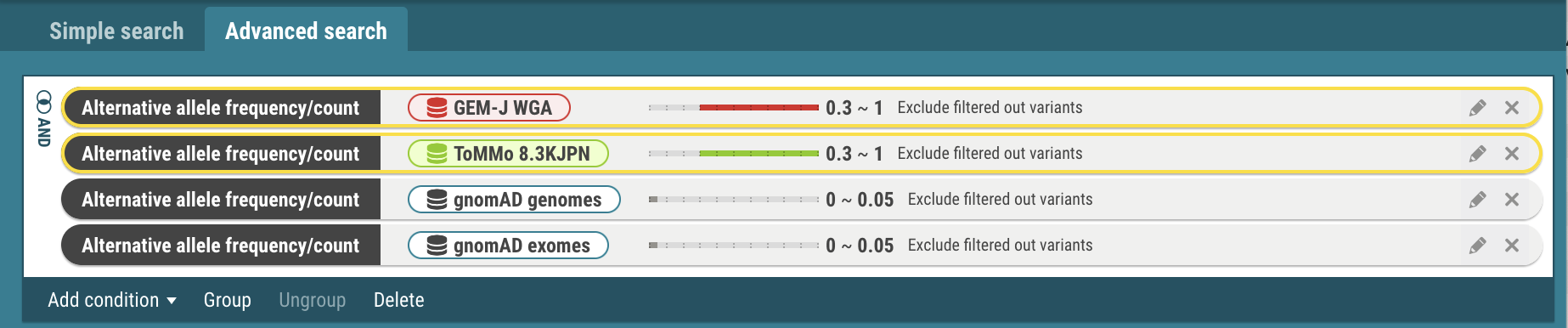
- 日本人集団(GEM-J WGAとToMMo 8.3KJPN)の検索条件をOR結合にします。
- GEM-J WGAとToMMo 8.3KJPNの検索条件をクリックして選択します。選択状態では黄枠で囲まれます。
- Groupメニューを選択します。選択した2つがOR条件になります。なお、AND条件に変更する場合は、左端のORアイコンをクリックします。
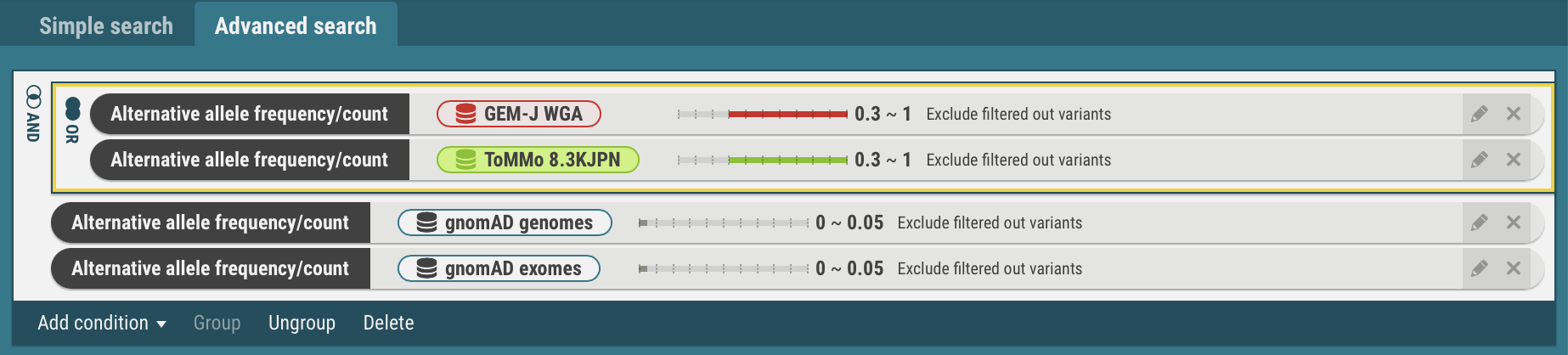
- 同様にgnomAD genomesとgnomAD exomesもOR条件にします。
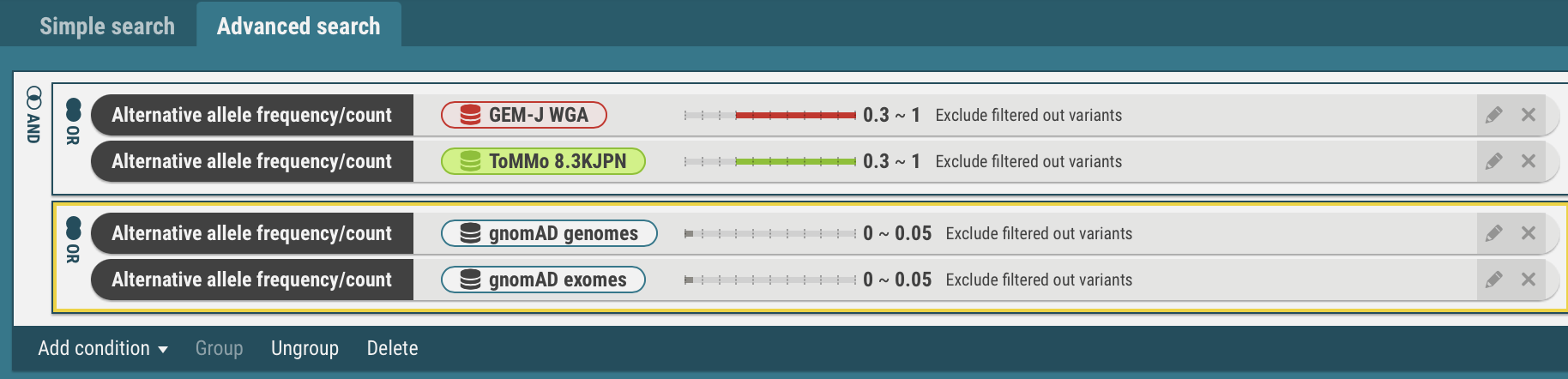
- GEM-J WGAとToMMo 8.3KJPNの検索条件をクリックして選択します。選択状態では黄枠で囲まれます。
AND
(freq(gnomAD genomes) <= 0.05) OR (freq(gnomAD exomes) <= 0.05))検索条件が変更される度に検索が自動的に実行され、条件に合致するバリアントの一覧が更新されます。
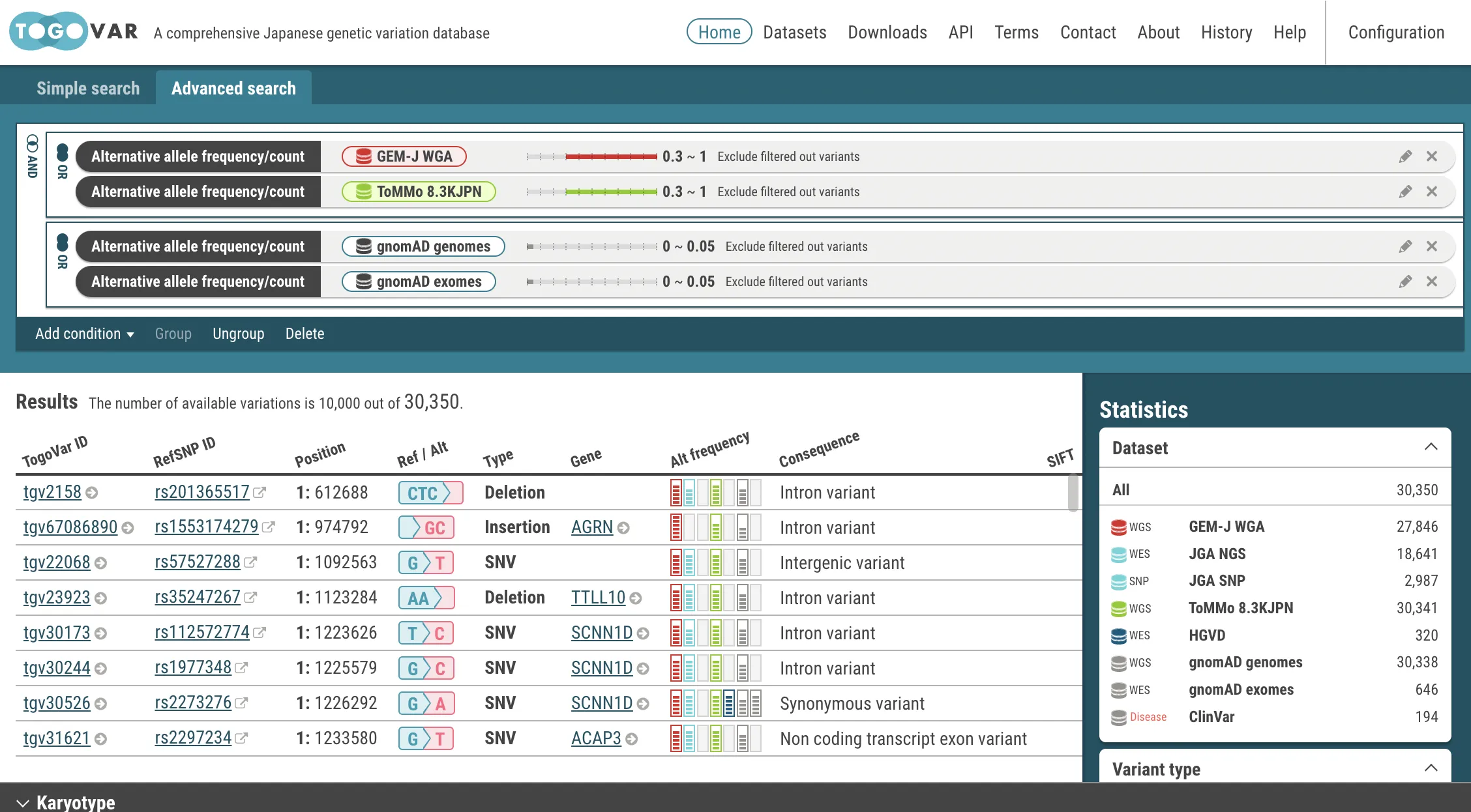 メニューの機能
メニューの機能- Add condition: 検索条件を追加します。
- Group: 選択された検索条件(黄枠表示)をOR条件で結合します。ORアイコンをクリックするとAND結合に変更できます。
- Ungroup:結合された検索条件を分離します。Groupとは反対の操作です。
- Delete:選択した 検索条件を削除します。
 :検索条件を編集します。
:検索条件を編集します。 :検索条件を削除します。
:検索条件を削除します。
GRCh37、GRCh38のどちらの位置でもバリアントを検索できますか。